Googlomics : Le cancer scruté par l’algorithme de Google
Une méthode dérivée du célèbre algorithme PageRank de Google et empruntant à la théorie quantique de la diffusion a été appliquée à un réseau de protéines et a permis de déceler des relations causales cachées entre certaines protéines. Cette méthode permet de mieux comprendre les liens entre les protéines, notamment comment ceux-ci sont modifiés dans le cas de pathologies.
Dans le corps humain, l’ensemble des protéines constitue un gigantesque réseau complexe, dont la structure évolue au cours du développement ou dans le cas de pathologies. Il existe plusieurs sous-réseaux comme ceux des protéines impliquées dans la transcription ou dans la signalisation cellulaire. Une fonction biologique donnée est déterminée par des liens de causalité entre les protéines (réseau dirigé), liens qui seront différents pour une autre fonction.
Une nouvelle méthode appelée Googlomics et mise au point conjointement par l’Institut UTINAM de Besançon (CNRS/UBFC), le Laboratoire de physique théorique de Toulouse (CNRS/UPS) et l’Unité cancer et génome de l’institut Curie de Paris (INSERM/PSL), analyse la structure des réseaux dirigés biologiques et permet de quantifier, de manière rigoureuse et rapide, les effets d’une protéine sur une autre en prenant en compte la structure globale du réseau biologique.
Le formalisme de la matrice de Google et de l’algorithme PageRank, dérivé des travaux de Sergei Brin et Lawrence Page, cofondateurs de Google, constitue la base mathématique du célèbre moteur de recherche. Il fournit la probabilité de visite de chaque page (PageRank) en analysant le processus aléatoire de visite d’un ensemble de pages web via les liens qui mènent (de façon dirigée) d’une page à une autre. Ce processus correspond à un cas particulier de chaîne de Markov.
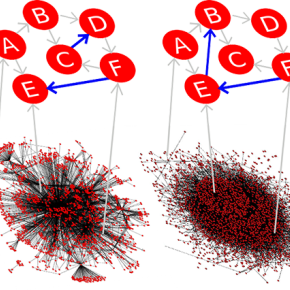
La méthode Googlomics consiste tout d’abord à transposer le réseau des pages web à un ensemble de protéines (les noeuds du réseau) ayant entre elles des relations causales (les liens du réseau). Elle utilise un développement de la matrice de Google fait par les chercheurs et appelé matrice de Google réduite, qui permet de calculer les liens effectifs pour un nombre restreint de nœuds sélectionnés, mais tout en considérant l’ensemble du réseau et donc en tenant compte de toutes les relations, directes et indirectes, entre ces nœuds.
Cette approche fournit ainsi un formalisme analytique efficace pour traiter numériquement des systèmes ayant des dizaines de milliers de nœuds et des centaines de milliers de liens. Elle s’inspire des méthodes de la théorie de la diffusion quantique développée par exemple en physique mésoscopique. Il y a en effet une analogie entre les interactions effectives existant entre les canaux ouverts de diffusion via l’ensemble des états de diffusion, et les relations effectives existant entre les nœuds sélectionnés via l’ensemble du réseau.
Les protéines d’intérêt pour la fonction biologique étudiée, par exemple en lien avec une pathologie, sont sélectionnées (ici une centaine environ) et la méthode Googlomics peut mettre en évidence de façon quantitative les relations causales cachées (via les liens indirects) entre elles, et en particulier les modifications induites par la pathologie.
Elle a ainsi permis de déceler le recâblage des connexions entre protéines dans plusieurs groupes de gènes associés au cancer et de quantifier les modifications du réseau transcriptionnel dans le cas de la leucémie myéloïde chronique.
Il y a fort à parier que cette méthode deviendra un outil important en biologie computationnelle.

© LPT (CNRS/UPS)
En savoir plus
Inferring hidden causal relations between pathway members using reduced Google matrix of directed biological networks
J. Lages, D. Shepelyansky, A. Zinovyev
PLoS ONE (2018), doi:10.1371/journal.pone.0190812
Informations complémentaires
Laboratoire de physique théorique (LPT, CNRS/Univ. Toulouse 3 Paul Sabatier)
Institut UTINAM (CNRS/Univ. Franche-Comté)
Unité Cancer et génome (Institut Curie/Inserm/Mines ParisTech/Univ. PSL)