Comment mieux estimer l'évolution du taux de reproduction de la Covid-19 ?
Une équipe de chercheurs interdisciplinaire a démontré que l'optimisation convexe constitue un outil mathématique et numérique efficace pour une estimation quotidienne de l’évolution du taux de reproduction de la Covid-19, au cours du temps et à travers les départements.
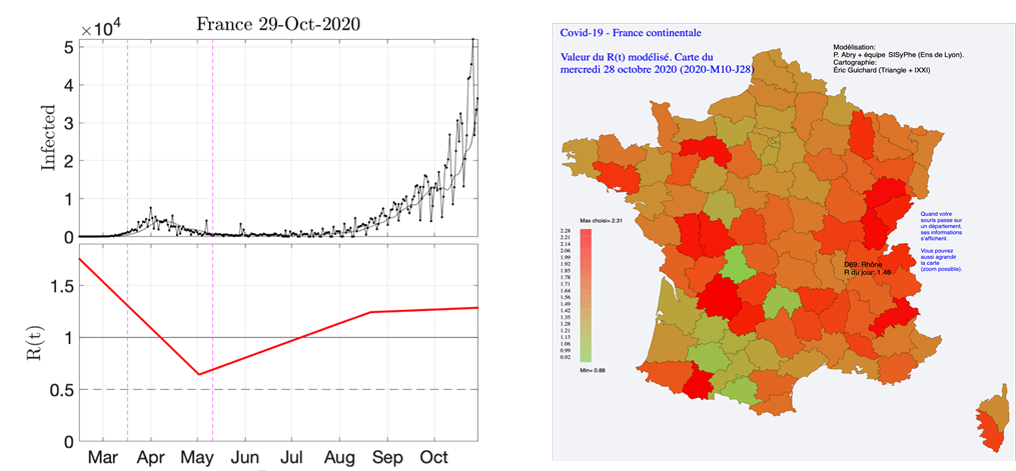
La surveillance de la propagation de la pandémie de Covid-19 constitue l’un des défis majeurs de la crise sanitaire mondiale. La mesure de l’évolution d’une pandémie se réalise théoriquement à l’aide de modèles épidémiologiques rendant compte d’interactions dans une population entre individus sains, exposés, malades, guéris ou décédés. L’utilisation pratique de ces modèles mathématiques implique cependant l’estimation, à partir des infections, guérisons et décès, d’un grand nombre de paramètres. La situation d’urgence créée par la pandémie Covid-19 s’est traduite par la difficulté de collecter des données épidémiologiques de qualité suffisante. Des versions plus opérationnelles de ces modèles ont été proposées, centrées sur un seul indicateur essentiel de l’évolution d’une pandémie : le taux de reproduction, qui rend compte du nombre probable de prochaines infections induites par chaque personne infectée. Une équipe interdisciplinaire rassemblant des spécialistes de traitement du signal et de l’information, des informaticiens, des physiciens, des géographes du Laboratoire de physique (LPENSL, CNRS/Université Claude Bernard Lyon 1/ENS Lyon), du Laboratoire d'informatique du parallélisme (LIP, CNRS/INRIA/ Université Claude Bernard Lyon 1/ENS Lyon) et du Laboratoire triangle (Triangle, CNRS/ENS Lyon/Université Lumière Lyon 2/Université Jean Monnet/Sciences Po Lyon), propose une approche originale reposant sur l’optimisation convexe, afin de réaliser une estimation quotidienne fiable de l’évolution du taux de reproduction au cours du temps et à travers différents territoires, le tout à partir de données imparfaites et dans un contexte d’urgence.
Le taux de reproduction se définit comme le rapport entre le nombre de nouvelles infections observées à une date donnée et une moyenne des nombres de nouvelles infections observées les jours précédents, cette moyenne étant pondérée par une fonction qui modélise la période de contamination d’une personne malade. Pour estimer ce taux de reproduction, les chercheurs ont transformé le modèle mathématique qui met en relation le nombre quotidien de nouvelles infections et le taux de reproduction en une fonction qui réalise un compromis entre le respect du modèle mathématique et un ensemble de contraintes que cet indicateur épidémiologique doit, par nature, respecter : être positif, évoluer lentement au cours du temps et varier faiblement entre territoires voisins. Néanmoins, la fonction est également construite pour prendre en compte de possibles ruptures dans cette évolution, dues par exemple à l’impact de politiques sanitaires. Les chercheurs ont ensuite élaboré l’algorithme, reposant sur des techniques numériques et avancées théoriques récentes en minimisation. Le minimum de cette fonction fournit l’estimation la plus robuste de l’évolution spatio-temporelle du taux de reproduction. Cet outil, mis en œuvre grâce aux données de Santé-Publique-France et du European Center for Disease Prevention and Control, permet de connaître en temps réel l'intensité de l'épidémie, et de mesurer l'impact des différentes mesures prises pour contrer l'épidémie, y compris de manière rétroactive. Enfin, il offre la possibilité de détecter une tendance quant à l'évolution du taux de reproduction. Il devient ainsi possible d'estimer quand il deviendra inférieur à 1, et donc, de savoir quand le nombre de nouvelles infections atteindra un maximum, puis en ajoutant une dizaine de jours, quand interviendra le pic des hospitalisations, etc., cela dans l'ensemble des départements français et dans plus de deux cents pays.
Référence
Spatial and temporal regularization to estimate COVID-19 reproduction number R(t): Promoting piecewise smoothness via convex optimization
Patrice Abry, Nelly Pustelnik, Stéphane Roux, Pablo Jensen, Patrick Flandrin, Rémi Gribonval, Charles-Gérard Lucas, Éric Guichard, Pierre Borgnat, Nicolas Garnier, PLoS ONE, le 20 août 2020
DOI : 10.1371/journal.pone.0237901
